Meeting Abstract
Autoren
- Bartlomiej Marzec - Georg-August-Universität, Göttingen, DE
- Philipp Weil - Universitätsmedizin Göttingen, Göttingen, DE
- Sara Yasemin Nussbeck - Universitätsmedizin Göttingen, Georg-August-Universität, Göttingen, DE
Einleitung und Fragestellung
Elektronische Laborbücher (ELN) ermöglichen die Nachnutzung von Forschungsdaten (FD) durch eine Annotation mit Metadaten (MD). Im Rahmen des Sonderforschungsbereiches (SFB)1002 an der Universitätsmedizin Göttingen wird ein ELN eingeführt. Um Forschern die strukturierte Dokumentation in ELNs zu ermöglichen, ist es notwendig Datenerfassungsformulare zu erstellen. Diese Formulare dienen sowohl der Annotation von FD mit MD, als auch der Abbildung von Abhängigkeiten zwischen Forschungsdatensätzen. Da Einträge in Laborbüchern die Grundlage für Veröffentlichungen darstellen, müssen die Empfehlungen zur Sicherung guter wissenschaftlicher Praxis eingehalten werden [1]. Diese impliziert die Verwendung definierter Standards für die nachhaltige Aufbewahrung der Primärdaten. Dazu wird von den Förderern die Speicherung der FD in nachnutzbarer Weise verlangt. Die zu verwendenden Standards sind im biomedizinischen Bereich zumeist in so genannten Minimum Information Checklists [2] zusammengefasst. Die Herausforderung dabei besteht darin, dass diese Standards um benutzerspezifische Anforderungen ergänzt werden müssen. Der Vorbereitungsprozess für die elektronische Dokumentation und Nachnutzbarkeit von FD soll hier am Beispiel von Phänotypisierungsdaten (Echokardiogramme (Echo) und Operationen (OP)) von Herzinsuffizienz am Mausmodell aufgezeigt werden. In Zusammenarbeit mit dem Institut für Zelluläre und Molekulare Immunologie in Göttingen wurde dieser generisch anzuwendende Prozess entwickelt.
Material und Methoden
Aus den Datenmanagementplänen des Digital Curation Center und der Checkliste des WissGrid-Projekts wurde ein Fragebogen für den Anwendungsfall extrahiert. Basierend auf diesem Fragebogen wurden Interviews mit den Laboranwendern durchgeführt. In Abhängigkeit des Interviewergebnisses wurde eine Literaturrecherche über existierende biomedizinische Metadatenstandards und -guidelines durchgeführt. Für die Ableitung der Beziehungen zwischen den Forschungsdatensätzen wurde eine Analyse der Arbeitsabläufe in Gesprächen mit den Forschern ermittelt.
Ergebnisse
Am Beispiel des SFB1002 in der Immunologie wurde an Hand der Interviews und Workflowanalysen deutlich, dass die strukturierte Annotation in Datenerfassungsformularen, die Erfassung von einzelnen Experimenten (z.B. Echo von operierten Mäusen) nicht ausreichend ist. Vielmehr müssen auch Versuchsaufbau (1. Prä-OP Echo 2. Operation, 3. Post-OP-Echo) –durchführung (Annotation des Experiments: z.B. Nadelstärke, Betäubungsmittel, Einstellung des Echokardiographen) und das einzelnen Versuchsobjekt (Maus) detailliert beschrieben werden. Da in der Literaturrecherche keine Standards für Echokardiographische Experimente zu finden waren, wurden die Templates auf Grundlage der von den Forschern gesammelten Daten und Allgemeinstandards wie Dublincore [3] erstellt. Teile der technischen Daten stammen aus dem Minimal Information for Mouse Phenotyping Procedures [4] und dem Minimum Information about a Flow Cytometry Experiment [5]. Aus diesem Beispiel lassen sich die einzelnen Arbeitsschritte für die Erstellung von Datenerfassungsformularen für ELNs ableiten:
- Erstellen eines reduzierten DMP-Fragenkatalog,
- Interviews und Workflowanalyse mit Experimentalwissenschaftlern des Fachgebiets.
- Literaturrecherche basierend auf den Interviews zur Ermittlung bestehender MD-Standards.
- Abgleich der Interviews mit den aus 3. ermittelten MD-Standards
- Strukturieren der Datenerfassungsformulare durch die Summe der aus 4. ermittelten MD und der in 2. durchgeführten Workflowanalyse.
Diskussion
Die erstellten Datenerfassungsformulare für Echos an operierten Mäusen müssen im Praxiseinsatz validiert und optimiert werden. Darüber hinaus wurde in den Interviews festgestellt, dass die Kommunikation zwischen Experimentalwissenschaftlern und IT zunächst eine gemeinsame Nomenklatur benötigt, da allein die Definition des Wortes „Experiment“ zu Missverständnissen führte. Aus den Ergebnissen der Interviews lässt sich auch ableiten, dass das Bewusstsein der Wissenschaftler gegenüber Metadatenstandards geschult werden muss.
Literatur
- Deutsche Forschungsgemeinschaft. Empfehlungen zur gesicherten Aufbewahrung und Bereitstellung digitaler Forschungsprimärdaten. Bonn: Wissenschaftliche Literaturversorgungs- und Informationssysteme; 2009.
- Taylor CF, et al. Promoting coherent minimum reporting guidelines for biological and biomedical investigations: the MIBBI project. Nat Biotechnol. 2008;26:889–96.
- ISO 15836 - Information and documentation -- The Dublin Core metadata element set 2009.
- Hancock JM, et al. Mouse Phenotype Database Integration Consortium: integration [corrected] of mouse phenome data resources. Mamm Genome. 2007;18:157–63.
- Lee JA, et al. MIFlowCyt: the minimum information about a Flow Cytometry Experiment. Cytometry A. 2008;73:926–30.
Vortragsfolien
-

Folie 1: ...
-

Folie 2: Hintegrund

Folie 3: Ziele der Formularentwicklung
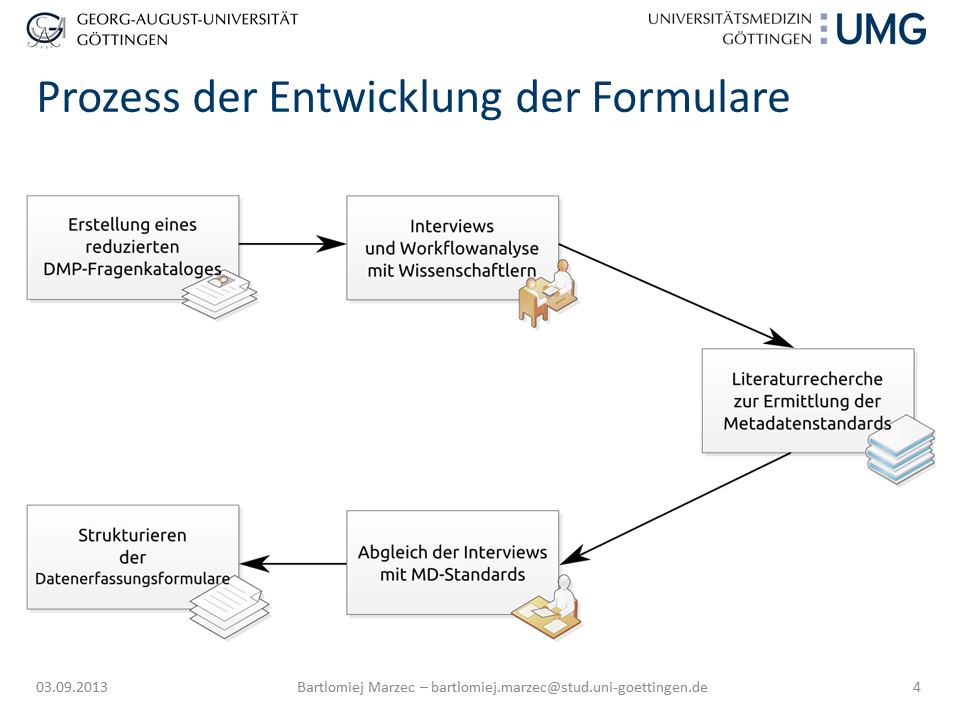
Folie 4: Prozess der Entwicklung der Formulare

Folie 5: Erstellung eines reduzierten DMP-Fragenkatalogs
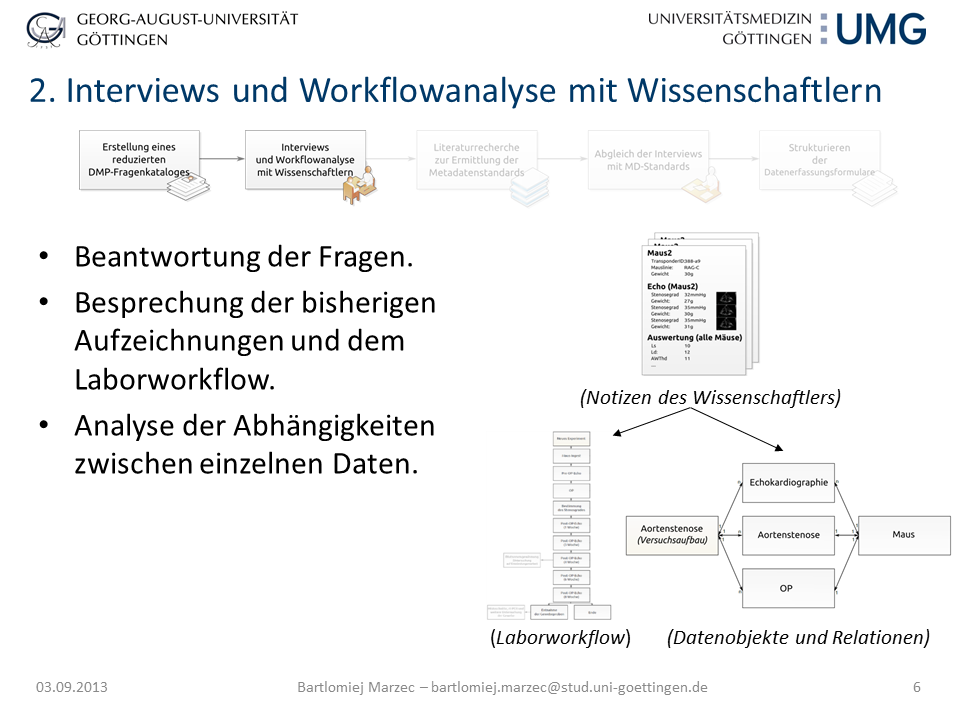
Folie 6: Interviews und Workflowanalyse mit Wissenschaftlern
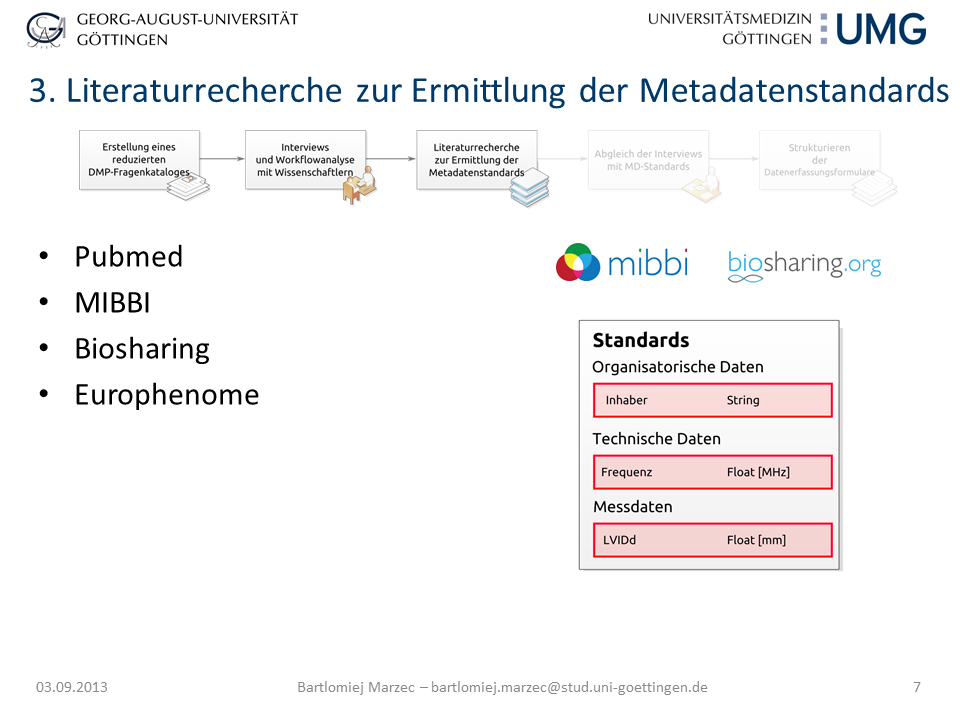
Folie 7: Literaturrecherche zur Ermittlung der Metadatenstandards
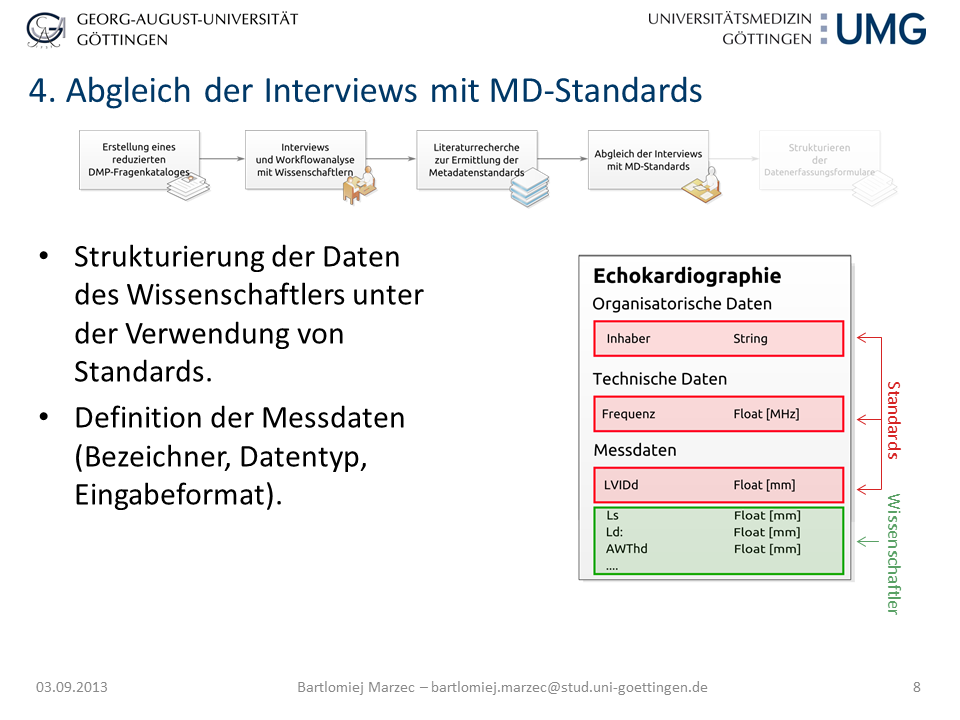
Folie 8: Abgleich der Interviews mit Metadatenstandards
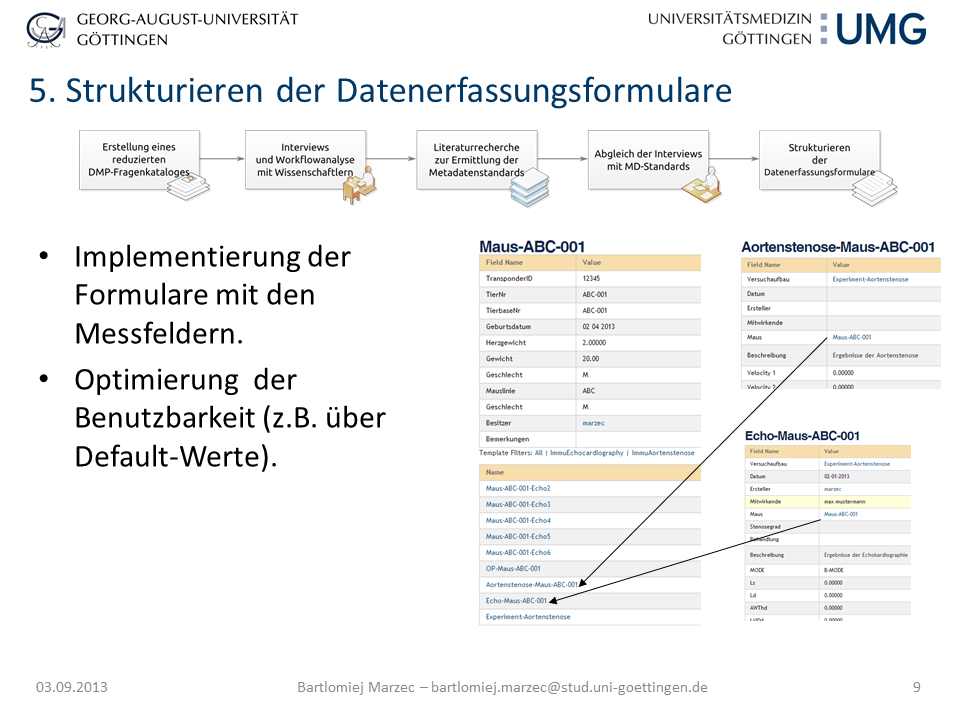
Folie 9: Strukturieren der Datenerfassungsformulare

Folie 10: Zusammenfassung

Folie 11: ...